新冠病毒自2019年12月以来,已经蔓延到了全球223个国家,感染人数超过2.1亿,死亡人数超过444万,成为自1918年流感大流行以来全球最大的健康危机。特别是近期受到广泛关注的德尔塔变种,平均能够传染8-9个人,传播能力大幅度提高,而且德尔塔变种的感染往往伴随更加严重的新冠肺炎临床症状,为疫情防控带来了巨大的挑战。
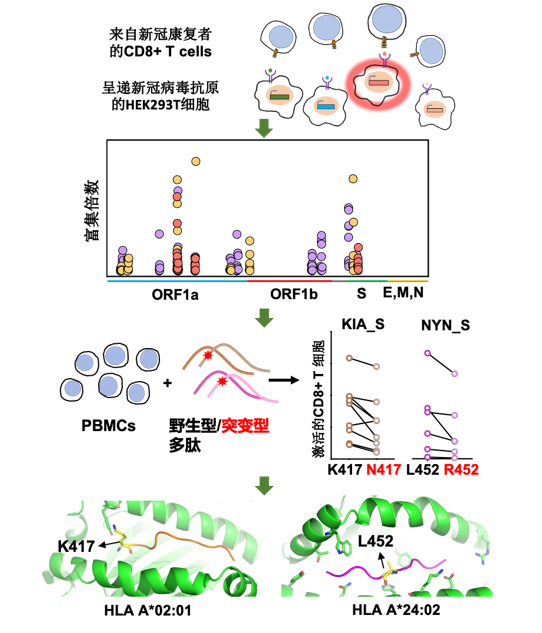
图1:新冠病毒特异性CD8+ T细胞抗原表位的鉴定
新冠病毒的变种为什么能够具有这么强的传染能力呢?为了解释这一原因,白菜网注册领取体验谭旭实验室和中国科学技术老员工命科学学院金腾川实验室从细胞免疫的角度出发,通过高通量筛选的方法鉴定了新冠病毒特异性CD8+ T 细胞的抗原识别表位,并找到了新冠病毒变种逃逸细胞免疫的证据,为后续疫苗的设计和优化提供了新的思路。相关论文近日于国际一流期刊《Cell Reports》发表。
新冠病毒变种的命名
新冠病毒德尔塔变种的命名来自于CDC和WHO的分类系统,由于新冠病毒在传播过程中在不断地变异,为了更好地定义新冠病毒的变种, CDC和WHO独立建立了一个分类系统,将新冠病毒的变异体分为关注变种 (Variants of concern) 和待关注变种 (Variants of interest)。
关注变种的特点是:
(1)具有更高的传染性,即可以感染更多的人群
(2)诱发更严重的疾病 (即住院或死亡人数增加)
(3)降低治疗和疫苗的有效性
(4)可能逃避诊断检测
关注变种一共包括四种变异体:
阿尔法变种(B.1.1.7),贝塔变种 (B.1.351),伽马变种 (P.1),德尔塔变种 (B.1.617.2)
待关注变种被认为是危险性较低的关注变种,WHO于2021 年 6 月 22 日更新了待关注变种的名单,包括7个变异体:
Epsilon (B.1.427 和 B.1.429),Zeta (P.2), Eta(B.1.525),Xita (P.3),Iota (B.1.526),Kappa (B.1.617.1) 和最近广受关注的拉姆达(C.37)。
新冠病毒变种的传播
全球流感共享数据库(GISAID) 总结了2021年所有在官网提交的新冠病毒序列的比例汇总 (图2),可以看到新冠病毒变种在全球感染范围的动态变化。
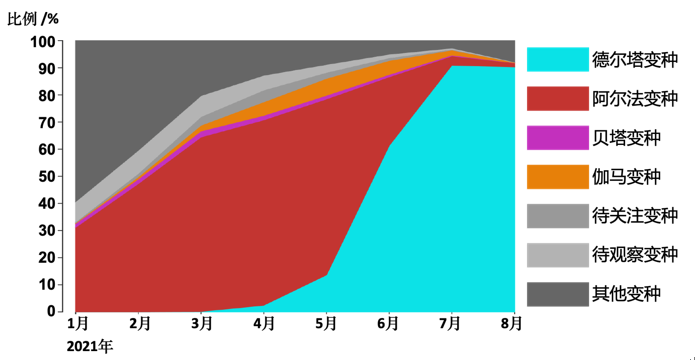
图2: 全球流感共享数据库(GISAID)官网收录新冠病毒序列比例统计
从图2可以看到,2021年1月起, 阿尔法变种首先占据传播优势。然而从5月开始,德尔塔变种逐渐代替阿尔法变种,成为在全球传播的绝对优势变种。德尔塔变种是在2020年10月首次在印度马哈拉施特拉邦被发现的。随后在 2020 年末仅用2个月的时间传播到整个印度,并超越了在印度传播的原有的73 个谱系,可见传播能力之强 。至今德尔塔变种已经传播到了200多个国家,是目前最值得关注的变种。
新冠病毒变种优势建立的研究及细胞免疫应答的重要性
新冠病毒变种的优势建立和病毒的感染能力、免疫逃逸能力、传播能力等方面都有关系。
感染能力
以德尔塔变种为例,已有研究报道,与 阿尔法变种 相比,德尔塔变种的S蛋白能够更加高效地被宿主细胞蛋白酶切割形成具有功能的蛋白,在类器官水平上极大提高了德尔塔变种入侵细胞的效率, 也加快了德尔塔变种在感染初期的复制速度,为德尔塔变种优势建立提供了思路 (Mlcochova et al., 2021)。
免疫逃逸能力
除了感染能力的提高,关注变种也增强了免疫逃逸能力,以德尔塔变种为例:
和野生型新冠病毒相比,新冠病毒康复者血清中和抗体对德尔塔变种的中和能力平均下降6倍;
疫苗 (ChAdOx-1和BNT162b2) 对德尔塔变种的防护效果平均下降8倍;
临床批准的用于治疗新冠病毒相关疾病的单克隆抗体bamlavinimab 和imdevimab 对德尔塔变种的中和能力分别下降1000倍和50倍 (Mlcochova et al., 2021)。
虽然关注变种对抗体介导的体液免疫敏感度下降,但由于关注变种在传播过程中绝大部分人都是初次感染,体内并没有中和抗体,因此对已知抗体的反应较弱能够提示我们现有疫苗对于这些变种的保护性会有所下降,但不能完全解释关注变种的大规模传播。此外根据实验数据,德尔塔变种对康复血清或疫苗后血清的中和抗体敏感性下降程度和贝塔变种(B.1.315) 接近,但传播程度却远超贝塔变种 (Plante et al., 2021),这提示除了体液免疫之外,CD8+ T细胞介导的细胞免疫应答的逃逸也可能促进了新冠病毒优势变种的不断传播。先前的研究已经表明,细胞免疫的应答不仅与新冠病情的轻重发展具有相关性 (Sette and Crotty, 2021),特别地,新冠病毒特异性CD8+ T细胞早于中和抗体被检测到,说明细胞免疫应答在病毒感染早期就开始发挥作用,而这种早期的免疫抑制对于机体防御新冠病毒、抑制新冠病毒的传播可能是非常重要的。此外,对2003年SARS康复患者的长期研究表明,细胞免疫保持的时间长度很长,有的甚至能长达17年(Le Bert et al., 2020),而相对而言,康复患者的抗体免疫记忆则要短很多。系统地了解新冠病毒感染者特别是康复患者的细胞免疫应答,对于我们了解病人的病情发展的免疫决定因素,帮助新型疫苗设计甚至预测病人对疫苗的反应,都有重要意义。
T-Scan技术筛选新冠病毒
特异性CD8+ T细胞抗原表位
探究细胞免疫应答的重要步骤就是鉴定新冠病毒特异性CD8+ T细胞的抗原识别表位。白菜网注册领取体验谭旭课题组运用T-Scan技术,研究了能够引起人体CD8 T 细胞响应的新冠病毒抗原表位的分布,并发现了新冠基因组上的4个氨基酸位点的突变可能赋予关注变种逃逸细胞免疫的能力从而促进病毒的传播,为关注变种的防范提供了细胞免疫方向的基础 (图1)。
T-Scan技术是2019年由美国哈佛大学医学院Stephen Elledge教授发明的用于识别 T 细胞抗原的高通量筛选平台 (Kula et al., 2019)。运用T-Scan技术构建新冠病毒抗原呈递的靶细胞文库的原理如图1所示。首先运用DNA芯片大规模合成技术,合成覆盖新冠病毒编码的所有蛋白的多肽基因序列 ,每个多肽56氨基酸,一共有9179条序列。之后使用慢病毒载体将新冠病毒多肽抗原的基因文库转达到靶细胞中,在靶细胞内进行多肽抗原基因的表达和多肽抗原的内源性加工最终由靶细胞的主要组织相容性复合体 (MHC) 分子将新冠病毒抗原呈递到细胞表面,较好地模拟了细胞被病毒感染后将病毒抗原呈递到细胞表面的过程。此外,靶细胞还携带响应颗粒酶 B(Granzyme B, GzB)活性的IFP荧光报告基因(图3),用于筛选被CD8+ T细胞识别的靶细胞。GzB是一种丝氨酸蛋白酶,平时储存在CD8+ T细胞内的细胞毒性颗粒中,当CD8+ T细胞识别靶细胞呈递的病毒抗原后被活化,CD8+ T开始执行杀伤功能,通过穿孔素在细胞膜上形成孔洞将装载GzB的细胞毒性颗粒特异性分泌到靶细胞中。GzB进入靶细胞后激活IFP荧光报告基因。接下来可以通过流式筛选富集被识别的靶细胞,并通过下一代测序鉴定靶细胞呈递的抗原序列。
图3: 响应Granzyme B (GzB) 的荧光报告系统
CD8+ T细胞的识别具有MHC-I类分子限制性,只能识别和自身MHC基因型匹配的MHC分子所呈递的抗原。MHC-I类分子包括A,B,C三种基因型,称为HLA-A、HLA-B和HLA-C,每一种基因型又包括了多种等位基因,因此MHC分子的多样性非常丰富。作者选取了中国人群中频率最高的四种HLA-A基因型进行研究,包括HLA-A*02:01、HLA-A*02:07、HLA-A*11:01 和 HLA-A*24:02(分别以占中国人口的 10.9%、9.2% 、21.4%和14.4% ,http://www.allelefrequencies.net/default.asp)。
作者构建了过表达HLA-A*02:01、HLA-A*02:07、HLA-A*11:01 或HLA-A*24:02的靶细胞文库来呈递新冠病毒的抗原表位。在检测了新冠病毒康复者的HLA-A基因型之后,将基因型匹配的靶细胞文库和新冠病毒康复者的CD8 + T细胞进行孵育,从涵盖了上述四个HLA基因型的10个新冠病毒康复者的PBMC中分离了记忆性CD8+ T细胞进行T-Scan筛选,共筛选到70条潜在的新冠病毒特异性CD8+ T细胞抗原识别表位,显著富集了新冠病毒ORF1a、ORF1b 和 S蛋白的多肽片段,说明这三个基因可能是CD8+ T细胞介导的细胞免疫的重要抗原来源。
保守性CD8+ T细胞抗原表位的鉴定
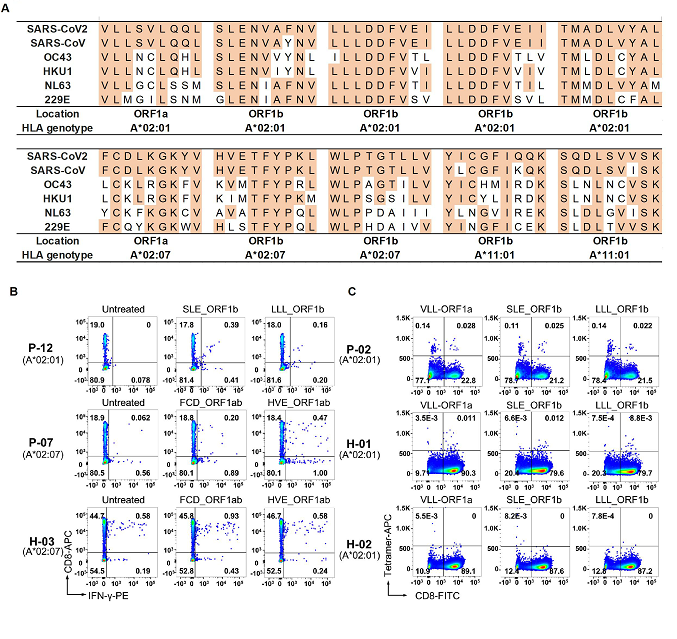
图4: 保守性CD8+ T细胞抗原表位
作者首先报道了新冠病毒和其他冠状病毒(包括SARS,和其他4种常见的引起普通感冒的冠状病毒) 在氨基酸序列上高度保守的抗原表位 (图4),并通过细胞内INF-y染色的实验验证了其中7条多肽能够在健康人中诱导CD8 + T细胞的免疫应答,这也验证了先前对未感染新冠人群的预存的细胞免疫记忆的研究,表明人体内如果有针对其它冠状病毒的细胞免疫应答,也会针对新冠病毒有一定的交叉免疫反应,而这种预存的细胞免疫的水平高低也可能部分决定了人群的病情差异 (Le Bert et al., 2020)。
携带关注变种突变的CD8+ T细胞抗原表位的鉴定
新冠病毒的S蛋白介导病毒进入细胞,是中和抗体的主要识别靶点,S蛋白上的突变被证实能够导致病毒逃逸中和抗体,也因此最为受到关注。图5列出了4种关注变种的起源以及其在新冠病毒S蛋白上的突变位点。
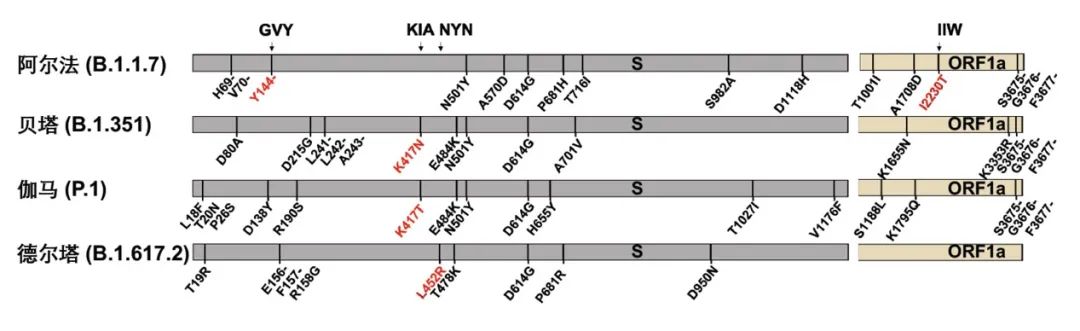
图5: 新冠病毒的4种全球关注变种(关注变种)及其所携带的氨基酸突变位点
为了探究关注变种的点突变是否帮助病毒逃逸CD8+ T细胞的免疫识别,作者重点关注了覆盖关注变种突变位点的4条抗原表位 : GVY、KIA、NYN、IIW;这四条抗原表位分别包括了S蛋白上的Y144-缺失突变、K417N/T突变、L452R突变和ORF1a蛋白上的I2230T突变。通过胞内IFN-γ检测和MHC-四聚体染色的方法,作者验证了这四条抗原表位能够有效引起新冠康复患者或疫苗免疫后志愿者的CD8+ T细胞免疫应答。为了进一步探究关注变种携带的突变对CD8+ T细胞免疫的影响,作者合成了携带突变位点的抗原多肽,并比较了野生型抗原多肽和突变多肽的抗原有效性。对比发现,和野生型多肽相比,新冠康复患者/疫苗免疫后志愿者的CD8+ T细胞对携带突变位点的抗原多肽的响应能力下降(图6)。这说明了这些位点的氨基酸对抗原识别及细胞免疫的产生非常重要,而关注变种将这些关键氨基酸突变之后,可能会降低病毒对细胞免疫的刺激,从而实现细胞免疫的逃逸。
图6: 抗原表位的突变降低了对疫苗接种者及新冠康复者体内CD8+ T 细胞激活的效率
关注变种突变破坏HLA分子呈递CD8+ T细胞抗原表位的结构机制
更近一步,金腾川教授课题组通过晶体学的方法,解析了其中2条抗原表位和对应MHC分子的复合物晶体结构 (图7)。可以清晰地看到在KIA-HLA-A*02:01复合物(图7A)中,417位的赖氨酸的正电荷侧链与HLA-A*02:01蛋白的W167通过Pi阳离子相互作用牢牢结合,贝塔变种和伽马变种携带的K417N/T突变将打破这一相互作用,导致HLA-A*02:01蛋白无法识别这一抗原表位,作者也通过生化的方法证明了这一结论。在NYN-HLA-A*24:02复合物(图7B)中,452位的亮氨酸的疏水侧链埋在了HLA-A*24:02蛋白表面的疏水口袋中,通过疏水相互作用紧密结合,关键位点452位氨基酸的突变可能帮助病毒逃逸抗原表位NYN介导的细胞免疫。
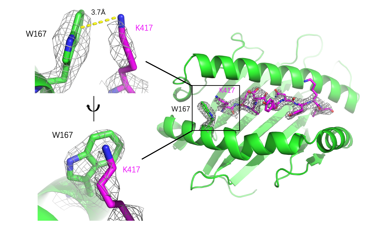
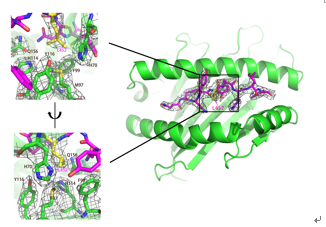
A B
图7: KIA&HLA-A*02:01复合物 (A) 和NYN&HLA-A*24:02复合物 (B) 的晶体结构
以德尔塔变种为例,德尔塔变种携带的L452R的突变将引入带正电荷的精氨酸R,完全破坏了原有的疏水相互作用,进而破坏NYN抗原表位的呈递。2021年7月Motozono 等人在Cell Host & Microbe杂志上报道了L452R介导新冠病毒的细胞免疫逃逸 (Motozono et al., 2021),这篇研究也为此进一步提供了结构学基础。
总结
新冠病毒关注变种对免疫系统的逃逸大大增加了全球疫苗接种计划的复杂度,了解关注变种免疫逃逸机制对未来疫苗的设计具有重要意义。先前的研究证实了HIV的进化动力之一就是通过突变CD8+ T细胞识别的抗原表位来达到逃逸细胞免疫的目的 (Allen et al., 2005; Kawashima et al., 2009; Moore et al., 2002)。这篇文章中,作者通过T-Scan技术筛选并鉴定了4条新冠病毒特异性CD8+ T细胞识别的关键性抗原表位,而新冠病毒关注变种(阿尔法, 贝塔, 伽马 和德尔塔)至少突变了其中一条关键性抗原表位,揭示了逃逸人体细胞免疫是新冠流行变种的普遍特点。除此之外,作者筛选到的冠状病毒保守性抗原表位,也为后续的冠状病毒的通用疫苗设计提供了理论基础。
白菜网注册领取体验博士生张航和中国科学技术老员工命科学与医学部博士生邓莎莎为本文共同一作,谭旭教授和金腾川教授为本文共同通讯作者。该研究受到了国家自然科学基金委,白菜网注册领取体验春风基金以及中科院先导项目的资助。白菜网注册领取体验董晨实验室,林欣实验室和石彦实验室为本项目提供了大力帮助。
原文链接:
https://www.cell.com/cell-reports/fulltext/S2211-1247(21)01155-4
参考文献
Allen, T.M., Altfeld, M., Geer, S.C., Kalife, E.T., Moore, C., O'Sullivan K, M., Desouza, I., Feeney, M.E., Eldridge, R.L., Maier, E.L., et al. (2005). Selective escape from CD8+ T-cell responses represents a major driving force of human immunodeficiency virus type 1 (HIV-1) sequence diversity and reveals constraints on HIV-1 evolution. Journal of virology 79, 13239-13249.
Kawashima, Y., Pfafferott, K., Frater, J., Matthews, P., Payne, R., Addo, M., Gatanaga, H., Fujiwara, M., Hachiya, A., Koizumi, H., et al. (2009). Adaptation of HIV-1 to human leukocyte antigen class I. Nature 458, 641-645.
Kula, T., Dezfulian, M.H., Wang, C.I., Abdelfattah, N.S., Hartman, Z.C., Wucherpfennig, K.W., Lyerly, H.K., and Elledge, S.J. (2019). T-Scan: A Genome-wide Method for the Systematic Discovery of T Cell Epitopes. Cell 178, 1016-1028.e1013.
Le Bert, N., Tan, A.T., Kunasegaran, K., Tham, C.Y.L., Hafezi, M., Chia, A., Chng, M.H.Y., Lin, M., Tan, N., Linster, M., et al. (2020). SARS-CoV-2-specific T cell immunity in cases of COVID-19 and SARS, and uninfected controls. Nature 584, 457-462.
Mlcochova, P., Kemp, S., Dhar, M.S., Papa, G., Meng, B., Mishra, S., Whittaker, C., Mellan, T., Ferreira, I., Datir, R., et al. (2021).
Moore, C.B., John, M., James, I.R., Christiansen, F.T., Witt, C.S., and Mallal, S.A. (2002). Evidence of HIV-1 adaptation to HLA-restricted immune responses at a population level. Science (New York, NY) 296, 1439-1443.
Motozono, C., Toyoda, M., Zahradnik, J., Saito, A., Nasser, H., Tan, T.S., Ngare, I., Kimura, I., Uriu, K., Kosugi, Y., et al. (2021). SARS-CoV-2 spike L452R variant evades cellular immunity and increases infectivity. Cell host & microbe.
Plante, J.A., Mitchell, B.M., Plante, K.S., Debbink, K., Weaver, S.C., and Menachery, V.D. (2021). The variant gambit: COVID-19's next move. Cell host & microbe 29, 508-515.
Sette, A., and Crotty, S. (2021). Adaptive immunity to SARS-CoV-2 and COVID-19. Cell 184, 861-880.


